Dentro del caos y la aleatoriedad más absolutos podemos encontrar
uno de los más sencillos y elegantes modelos matemáticos jamás
construido en la historia de la física teórica. Hoy analizaremos la
solución el enorme reto al que se enfrentaba la física de
finales del siglo XIX: explicar qué cosa era eso que llamamos
temperatura. Espero que os resulte amena esta introducción con animaciones y simulaciones, de las aprovecho para liberar el código fuente.
1. Jugando al billar
A cierto galo irreductible lo único que le asustaba en el mundo era que
el cielo se cayera un día sobre su cabeza, mucho más que unos cuantos
romanos de pacotilla.
Parece una pregunta tonta, pero ¿realmente has pensado alguna vez por qué no se cae el cielo? El
aire que nos rodea, la atmósfera de nuestro planeta, llega
aproximadamente hasta unos 100km de altura y está
formada, principalmente, por moléculas de nitrógeno y oxígeno que
sienten toda la fuerza de la gravedad tirando hacia abajo al igual que
la sentimos nosotros.
 |
| Esta preciosa puesta de sol, fotografiada desde la estación espacial ISS, revela perfectamente los pocos kilómetros de atmósfera que nos envuelven (fuente). |
Si un aciago día todas esas moléculas de la atmósfera "cayeran" al suelo formarían una capa sólida de apenas unos 13 metros de altura. Así que, ¿qué es lo que las sostiene "volando" a kilómetros de altura en forma gaseosa e impide que caigan?
Pues sorprendentemente, el puro y duro azar. Esas moléculas que pululan a nuestro alrededor a unos 2,000 km/h sí que están siempre intentando caer, pero una y otra vez se encuentran las unas con las otras y chocan, saliendo "rebotadas". Sabiendo que más del 99,9% del aire realmente es espacio vacío,
siendo el insignificante resto lo que ocupan las moléculas (que, a su
vez, internamente también están bastante huecas), resulta sobrecogedor
pensar en la astronómica cantidad de choques que deben ocurrir cada
segundo a nuestro alrededor para impedir que "el cielo se caiga".
Veamos un poco más de estos choques, ya que son la clave
que explicará qué es la temperatura. Nos bastará imaginarnos las
moléculas del aire como si fueran canicas, pequeñas esferas que vuelan
prácticamente libres en líneas rectas [nota 1]. Estamos acostumbrados a pensar que si algo atraviesa el aire a toda leche sufriría un rozamiento importante y se iría frenando. Pero eso no les ocurre a estas "canicas" ya que solamente existe el vacío y ellas, ¡así que no hay nada con lo que puedan "rozar"!
Por tanto lo único que puede interferir en el movimiento rectilíneo de
una de estas moléculas es... otra. Y la forma en que interaccionan se
pueden definir en palabras muy sencillas: se pegan porrazos de lo lindo unas contra otras.
Piensa que aún yendo a miles de km/h no se rompen. No es que sean
indestructibles, pero hace falta bastante más velocidad para partirlas.
Exceptuando la velocidad de los choques, un símil perfecto a estas colisiones es cuando en una mesa de billar la bola blanca choca contra otra que permanecía estática: la energía que llevaba la bola móvil se divide entre las dos, de una forma que asegura que la energía del movimiento (llamada energía cinética) antes y después del choque se mantiene constante.
 |
| Choque perfectamente elástico de dos esferas ideales. |
Vamos, aquello de que "la energía no se crea ni se destruye, sino que sólo se transforma". A este tipo de colisión se llama choque elástico porque
no se pierde nada de energía en deformaciones permanentes, y es el tipo
de choque que ocurre entre nuestras moléculas-canicas [nota 2]. Como curiosidad, decir que tanto en las moléculas como en las bolas de billar, esa fuerza tan poderosa que no deja que dos cuerpos sólidos se atraviesen mutuamente no es más que la repulsión eléctrica de
las cargas negativas que tiene la materia ("cargas de igual tipo se
repelen"). Brian Greene ilustró esto muy visualmente en el documental de
su libro "El universo elegante" (aquí subieron el vídeo).
2. Resumiendo información en un sólo número
Llegamos por fin a la temperatura: lo que llamamos temperatura es realmente un numerito que indica "cuánto se mueven" en total, todas las moléculas de un objeto o de un volumen de un gas como el aire.
¿Cómo es posible que un único número resuma el estado de trillones de moléculas? Aquí entra en juego el elegante resultado matemático que mencioné al empezar y que viene de mano de los choques entre moléculas.
Sabemos que la energía total de dos objetos que colisionan
elásticamente se mantiene constante, aunque lógicamente antes y después
del choque cada uno tenga velocidades (y por tanto energías) distintas.
Pero la suma, la energía total, no se altera.
Esto mismo se puede extender al choque de muchos objetos: si
una única molécula en movimiento entra en una caja cerrada donde hay
otras docenas de moléculas inicialmente en reposo, la energía de
movimiento (cinética) de la primera se repartirá entre todas las demás. Mejor verlo que imaginarlo: pulsa el play para ver entrar la molécula por la izquierda. Al igual que en el resto de vídeos, recomiendo ponerlo en alta resolución.
¿Cuál será la velocidad final de cada una de las bolitas? Nadie
puede decirlo. Fíjate que dependiendo del ángulo de los choques las
velocidades tras cada choque no dejan de variar, pudiendo aumentar o
disminuir, o incluso detenerse del todo.
Vamos, que la velocidad de una molécula en concreto depende de una forma
compleja de sus choques y de las velocidades de todas las demás, algo
absolutamente inabordable si pretendemos calcularlo...¡hasta que la estadística viene a nuestro rescate (pero no del bancario, sino del de verdad)!
Uno de los más grandes resultados de la matemática estadística nos dice
que si algo (lo que sea, p.ej. la velocidad de una molécula) viene de la
combinación de un gran número de sucesos aleatorios independientes (los
que sean, p.ej. direcciones de choques), entonces ese algo tendrá
valores aleatorios, al azar, pero siguiendo un patrón muy preciso llamado distribución Gaussiana. Esto se llama el teorema del límite central.
Si lo de "sigue una distribución Gaussiana" te parece chino mandarín,
esta animación te ayudará a entender qué quiere decir. Imagina que mides
miles de resultados de un experimento, el que sea, que contiene una
parte de azar, o de incertidumbre o un error. La cuestión es que cada
vez te sale un número distinto: un -1, un 2, un -0,5, etc. La línea negra del dibujo representa cada uno de esos resultados,
por eso va dando saltos al azar. Para hacernos una idea de cuántas
veces sale "casi -1", o "casi 2", etc. vamos contando las veces que el
resultado cae en una serie de divisiones horizontales, y eso son las barras azules que crecen cada vez que la línea negra las toca. La forma que acaban teniendo las barras azules se llama función densidad de probabilidad y a pesar de ser algo basado en el azar, siempre acabará teniendo una forma concreta y bien definida para cada experimento. La que ves abajo es precisamente una campana de Gauss, la misma distribución Gaussiana que nos apareció antes.
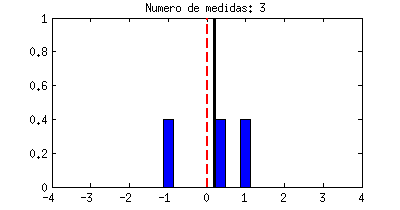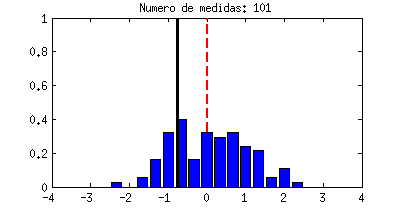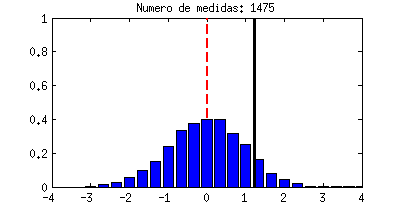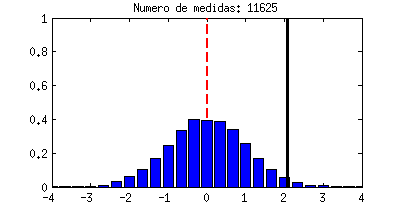 |
| Ejemplo de una variable aleatoria con distribución Gaussiana. |
Pues bien: lo que llamamos
temperatura no es más que un reflejo de estas campanas de Gauss y del
Teorema que dice que muchas variables aleatorias acaban siendo
Gaussianas. No importa por qué medio calentemos un gas, si disparándole
moléculas rápidas como en la simulación de arriba o cualquier otro
método, al final lo que hacemos es aumentar su energía cinética total.
Tras un tiempo, esa energía total acabará distribuyéndose entre todas las moléculas de forma que las velocidades en las direcciones arriba/abajo, izquierda/derecha y adelante/atrás serán todas distribuciones Gaussianas. Al mezclar las velocidades en las tres dimensiones
del espacio acaba saliendo que la velocidad tridimensional ya no es
Gaussiana, sino una combinación de ellas de una determinada forma que
los matemáticos llaman distribución chi y los físicos distribución de Maxwell-Boltzmann.
Pero ambas son la misma cosa y lo bello del asunto es que en el
fondo... ¡se reducen a combinaciones de sencillas Gaussianas! Así es
como existe una relación inequívoca entre la energía total de un gas
(que llamamos temperatura) y las velocidades de todas sus moléculas una
vez se ha alcanzado el equilibrio, dado por esas dos distribuciones.
Lea el artìculo completo en:




